covid19_ua
Аналіз "диванного експерта" відкритих даних по захворюваності на COVID19 в Україні
Project maintained by vityok Hosted on GitHub Pages — Theme by mattgraham
Динаміка поширення коронавірусної інфекції по областях
Нещодавно сайт “Тексти” поширив чудову статтю «Україна проти COVID-19: ні поразки, ні перемоги. Ми у групі країн, які найгірше справляються з пандемією» з цікавим графіком, на якому було зображено динаміку поширення коронавірусної хвороби в різних країнах. Добова кількість виявлених випадків була нормалізована для кожної країни, а тому графік дає можливість подивитись саме на динаміку, а не на масштаб епідемії у кожній окремій країні.
Нижче буде побудовано чимось схожий графік динаміки поширення коронавірусної інфекції COVID-19 в Україні, в розрізі областей, але без нормалізації. Аналі зроблений в системі R (прочитати більше у Вікі).
Також у Вікі можна дізнатись більше про перебіг епідемії в Україні.
Всі вихідні дані можна знайти в репозиторії covid19_ua.
Підготовка даних
Побудуємо графіки поширення коронавірусної інфекції COVID-19 (спричиненої вірусом SARS-CoV-2) по областях. Для цього скористаємось бібліотеками функцій з колекції tidyverse, зокрема ggplot2 для створення графіків, readr для зчитування, та dplyr для підготовки.
library(tidyverse)
library(ggridges)
library(slider)
Якщо системні налаштування локалі не співпадають з бажаними, їх можна змінити навіть тоді, коли сеанс роботи в R розпочато, для цього знадобиться функція Sys.setlocale:
Sys.setlocale(category="LC_ALL",locale="uk_UA.utf8" )
Тепер вже можна починати роботу. Зчитаємо із використанням функції read_csv бібліотеки tidyverse а не стандартної read.csv. Таким чином отримаємо дані одразу в структурі даних, зручній для подальшої роботи.
Як джерело даних можна вказати файл у локальній файловій системі:
area_dyn <- read_csv('../covid19_by_area_type_hosp_dynamics.csv')
Або ж завантажити найсвіжішу копію безпосередньо з серверів GitHub:
area_dyn <- read_csv('https://raw.github.com/VasiaPiven/covid19_ua/master/covid19_by_area_type_hosp_dynamics.csv')
Функція read_csv, окрім власне зчитування самої таблиці, іще й намагається правильно визначити типи даних, що зберігаються в кожному зі ствопчиків. І хоча розробники радять вказувати типи даних для кожного стовпчика явним чином, в нашому випадку це може бути зайвим, адже функція вірно визначає всі типи даних, навіть Date для стовпчика zvit_date.
head(area_dyn)
## # A tibble: 6 x 12
## zvit_date registration_ar… is_required_hos… person_gender person_age_group
## <date> <chr> <chr> <chr> <chr>
## 1 2020-11-08 Вінницька Ні Жіноча 0-9
## 2 2020-11-08 Вінницька Ні Жіноча 10-19
## 3 2020-11-08 Вінницька Ні Жіноча 10-19
## 4 2020-11-08 Вінницька Ні Жіноча 10-19
## 5 2020-11-08 Вінницька Ні Жіноча 20-29
## 6 2020-11-08 Вінницька Ні Жіноча 20-29
## # … with 7 more variables: add_conditions <chr>, is_medical_worker <chr>,
## # new_susp <dbl>, new_confirm <dbl>, active_confirm <dbl>, new_death <dbl>,
## # new_recover <dbl>
Таблиця в файлі covid19_by_area_type_hosp_dynamics.csv має доволі велику кількість стовпчиків, перелічемо їх для зручності подальшого використання в скрипті:
names(area_dyn)
## [1] "zvit_date" "registration_area"
## [3] "is_required_hospitalization" "person_gender"
## [5] "person_age_group" "add_conditions"
## [7] "is_medical_worker" "new_susp"
## [9] "new_confirm" "active_confirm"
## [11] "new_death" "new_recover"
Зараз нам цікаві лише кілька:
zvit_date, датаregistration_area, назва області або місто Київnew_susp, кількість підозр зареєстрованих протягом цього дняnew_confirm, кількість підтверджених випадків захворювання протягом дняnew_death, кількість зареєстрованих смертей протягом дня
Наступним кроком підрахуємо сумарні значення цікавих нам показників, що припадають на певний регіон протягом доби. Бібліотека tidyverse дає можливість зробити це функціями, що ззовні нагадують відповідні конструкції в SQL:
daily_area_reg_dyn <- area_dyn %>%
select(zvit_date, registration_area, new_susp, new_confirm, new_death) %>%
mutate(registration_area=factor(case_when(
registration_area == "м. Київ" ~ "м. Київ",
TRUE ~ stringr::str_to_title(registration_area)))) %>%
group_by(zvit_date, registration_area) %>%
summarise(new_susp = sum(new_susp),
new_confirm = sum(new_confirm),
new_death = sum(new_death))
## `summarise()` regrouping output by 'zvit_date' (override with `.groups` argument)
Нарешті, все готово для створення графіка.
Огляд
Та почнемо з побудови узагальнених даних для всієї країни. Для цього також знадобиться підрахунок сум, але в розрізі лише дат, без урахування регіонів:
daily_sum_dyn <- daily_area_reg_dyn %>%
group_by(zvit_date) %>%
summarise(new_susp = sum(new_susp),
new_confirm = sum(new_confirm),
new_death = sum(new_death)) %>%
filter(zvit_date > as.Date('2020-04-01'))
## `summarise()` ungrouping output (override with `.groups` argument)
ggplot(daily_sum_dyn) +
geom_line(aes(zvit_date, new_susp, colour='orange')) +
geom_line(aes(zvit_date, new_confirm, colour='blue')) +
geom_line(aes(zvit_date, new_death, colour='red')) +
scale_color_discrete(name="",
labels=c("Підтверджені",
"Підозри",
"Летальні")) +
theme_light() +
theme(legend.position = "bottom") +
labs(title="Загальний огляд перебігу епідемії COVID-19",
x = "Дата",
y = "Випадків за день",
caption = "Дані: ЦГЗ та НСЗУ")

Зробімо іще одне невеличке дослідження: зобразимо нормалізовані значення цих показників на гафіку.
max_susp <- max(daily_sum_dyn$new_susp)
max_confirm <- max(daily_sum_dyn$new_confirm)
max_death <- max(daily_sum_dyn$new_death)
daily_norm_dyn <- daily_sum_dyn %>%
mutate(new_susp = new_susp / max_susp,
new_confirm = new_confirm / max_confirm,
new_death = new_death / max_death) %>%
mutate(new_susp_7ma = slide_dbl(new_susp, mean, .size = 7, .align = "center"),
new_confirm_7ma = slide_dbl(new_confirm, mean, .size = 7, .align = "center"),
new_death_7ma = slide_dbl(new_death, mean, .size = 7, .align = "center"))
library(gridExtra)
##
## Attaching package: 'gridExtra'
## The following object is masked from 'package:dplyr':
##
## combine
library(grid)
daily_norm_dyn_confirm_death <- daily_norm_dyn %>%
select(zvit_date, new_confirm_7ma, new_death_7ma) %>%
pivot_longer(cols=contains('new_'),
names_to='metric',
values_to='val')
daily_norm_dyn_confirm_susp <- daily_norm_dyn %>%
select(zvit_date, new_confirm_7ma, new_susp_7ma) %>%
pivot_longer(cols=contains('new_'),
names_to='metric',
values_to='val')
breaks_metric <- c('new_confirm_7ma', 'new_susp_7ma', 'new_death_7ma')
labels_metric <- c('Підтверджених', 'Підозр', 'Летальних')
confirm_death_plot <- ggplot(daily_norm_dyn_confirm_death, aes(zvit_date, val)) +
geom_line(aes(color = metric)) +
theme_light() +
scale_color_discrete(breaks=breaks_metric, labels=labels_metric) +
theme(legend.position = "bottom") +
labs(title="Підтверджених та летальних",
x = "",
y = "Випадків за день",
caption = "")
confirm_susp_plot <- ggplot(daily_norm_dyn_confirm_susp, aes(zvit_date, val)) +
geom_line(aes(color = metric)) +
theme_light() +
scale_color_discrete(breaks=breaks_metric, labels=labels_metric) +
theme(legend.position = "bottom") +
labs(title="Підозр та підтверджених",
x = "Дата",
y = "Випадків за день",
caption = "Значення показників нормалізовано за максимальним. Дані: ЦГЗ та НСЗУ")
grid.arrange(confirm_death_plot, confirm_susp_plot, ncol = 1)

Підозри на захворювання COVID-19
Слід зазначити, що інформація в розрізі днів агрегована безпосередньо за датою реєстрації підозр, даті тестування/одужання/смерті та не залежить від дати, коли ці дані було оприлюднено.
Далі буде три однотипних графіка, тому детально розгляньмо будову лише першого: динаміка нових підозр на захворювання на COVID-19.
Оскільки графік має єдине джерело даних та однакові параметри вісей, це можна вказати одразу в конструкторі об’єкта ggplot:
plot_susp <- ggplot(daily_area_reg_dyn, aes(x=zvit_date, y=new_susp))
Далі до графіка слід додати визначення графічних об’єктів, що будуть на ньому зображені, в нашому випадку, це geom_line:
plot_susp <- plot_susp + geom_line()
Оскільки ми прагнемо створити окремий графік для кожної окремої області та міста Києва (стовпчик registration_area), слід додати відповідну інструкцію до графіка: facet_wrap робить саме це, тут слід вказати стовпчик, в якому знаходиться критерій для поділу, та кількість рядків або стовпчиків, у яких буде розташовано графіки.
plot_susp <- plot_susp + facet_wrap(vars(registration_area), ncol = 4)
GGplot2 має зручний механізм налаштування декорацій, який дає можливість як детальних підлаштувань під конкретні потреби, так і набори вже наперед заданих параметрів, так званих “тем”. Оберемо “легку” тему:
plot_susp <- plot_susp + theme_light()
Ну і нарешті визначимо такі атрибути графіка, як його назву, підпис, назви вісей, тощо:
plot_susp <- plot_susp + labs(title="Підозри на захворювання COVID-19",
x = "Дата",
y = "Підозр на день",
caption = "Дані: ЦГЗ та НСЗУ")
plot_susp
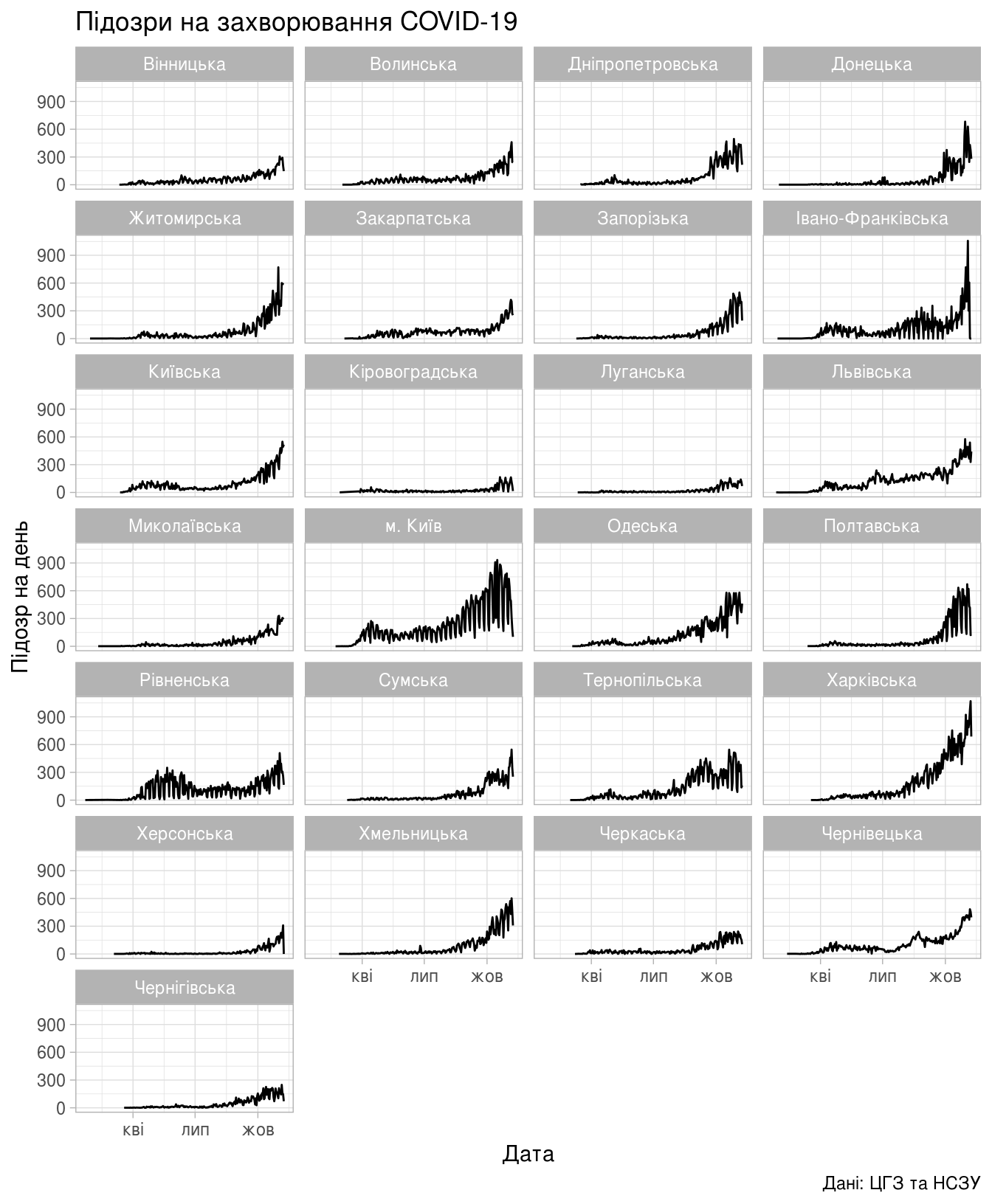
Результат.
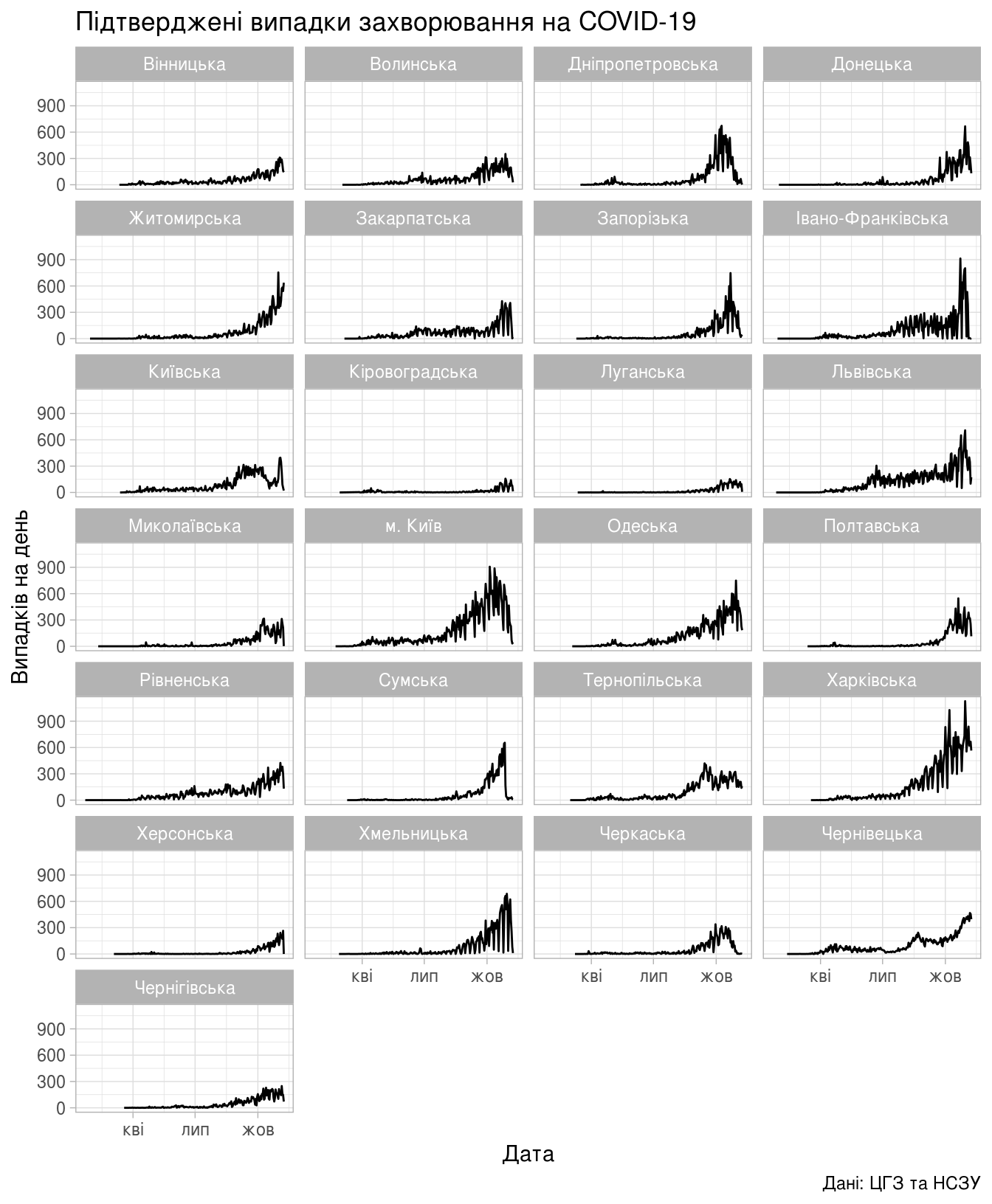
Підтверджені випадки захворювання на COVID-19
Динаміка лабораторно підтверджених випадків захворювань на COVID-19.
(ggplot(daily_area_reg_dyn, aes(x=zvit_date, y=new_confirm))
+ geom_line()
+ facet_wrap(vars(registration_area), ncol = 4)
+ theme_light()
+ labs(title="Підтверджені випадки захворювання на COVID-19",
x = "Дата",
y = "Випадків на день",
caption = "Дані: ЦГЗ та НСЗУ"))
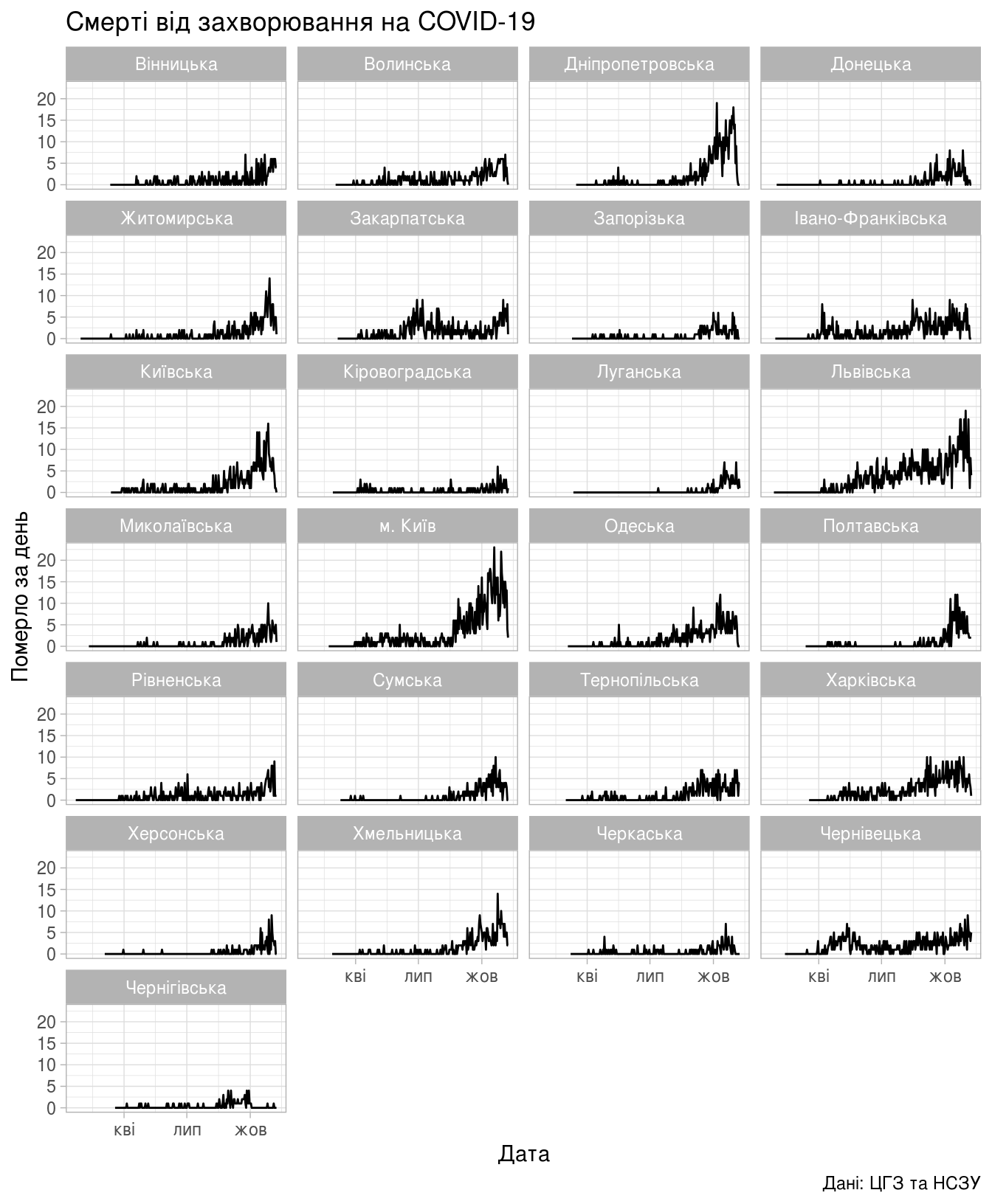
Смерті від захворювання на COVID-19
Динаміка летальних випадків у пацієнтів, хворих на COVID-19.
(ggplot(daily_area_reg_dyn, aes(x=zvit_date, y=new_death))
+ geom_line()
+ facet_wrap(vars(registration_area), ncol = 4)
+ theme_light()
+ labs(title="Смерті від захворювання на COVID-19",
x = "Дата",
y = "Померло за день",
caption = "Дані: ЦГЗ та НСЗУ"))
Інші варіанти
Всі області одразу (тут іще треба поліпшувати) із використанням бібліотеки ggridges (кількість летальних випадків):
(ggplot(daily_area_reg_dyn,
aes(x=zvit_date, y=registration_area,
height=new_death,
group=registration_area))
+ geom_ridgeline()
+ theme_ridges()
+ xlab("Дата")
+ ylab("Летальних випадків"))
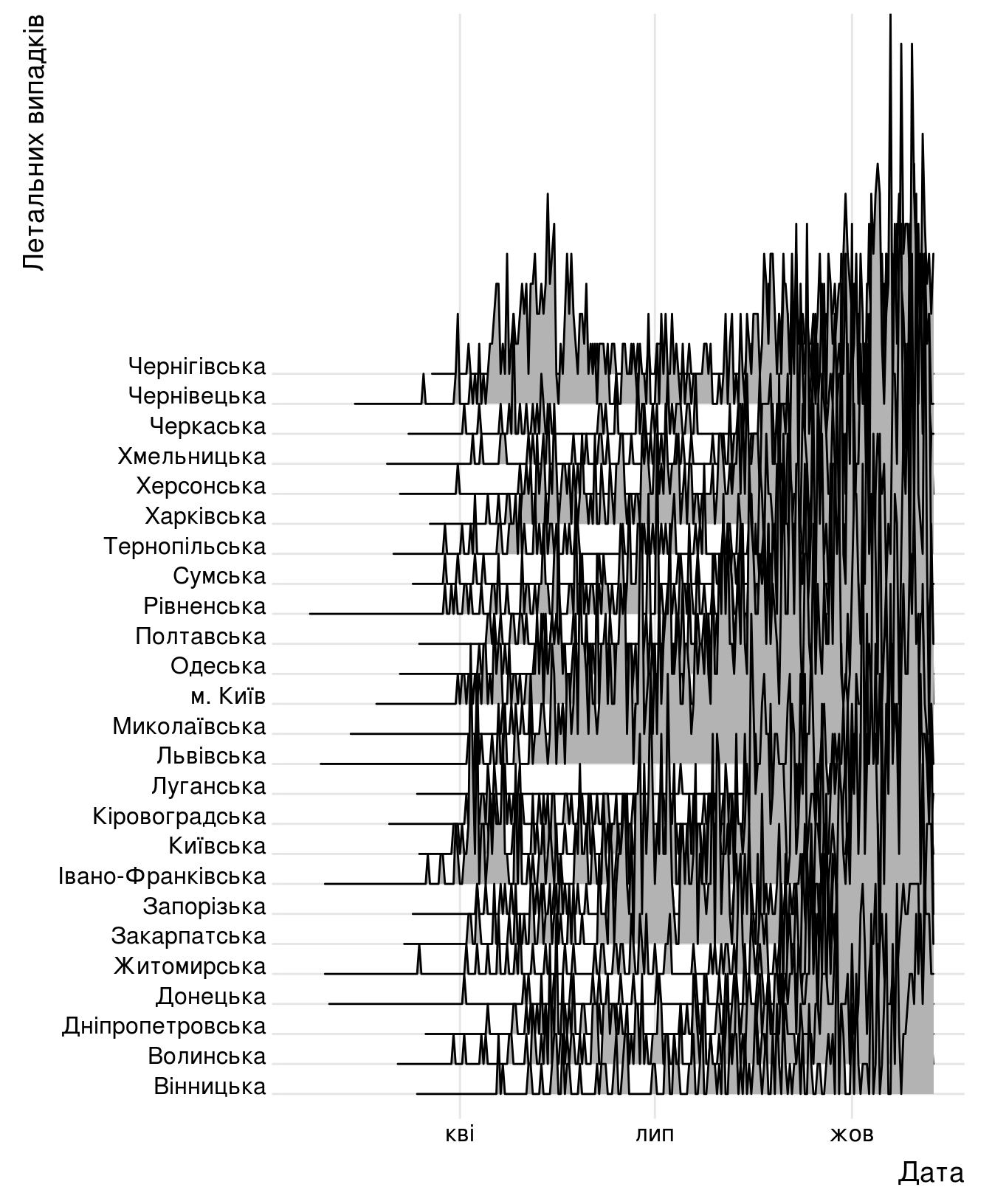
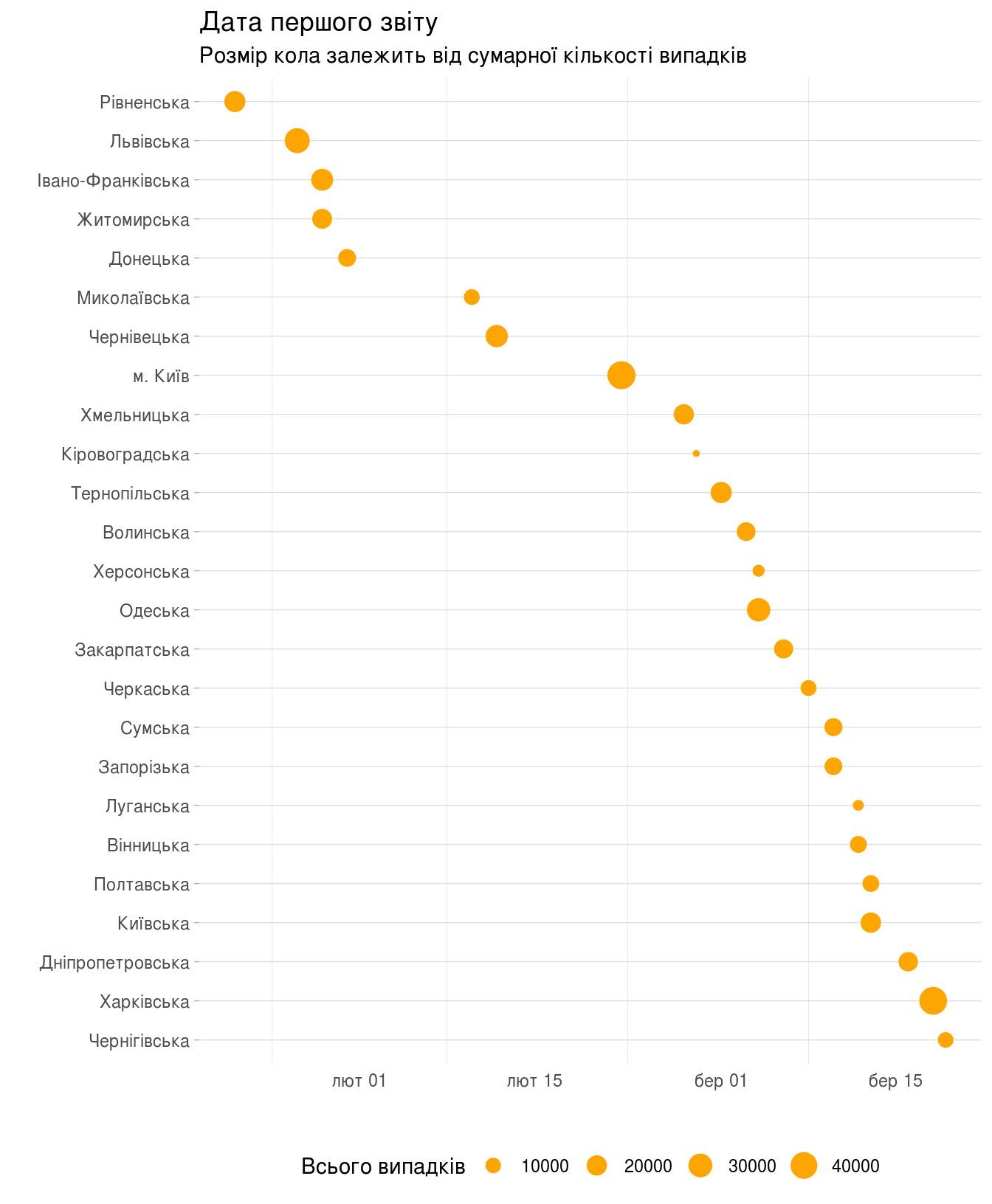
Коли був зареєстрований перший випадок в кожній області.
area_first <- area_dyn %>%
select(zvit_date, registration_area, new_confirm) %>%
mutate(registration_area=factor(case_when(
registration_area == "м. Київ" ~ "м. Київ",
TRUE ~ stringr::str_to_title(registration_area)))) %>%
group_by(registration_area) %>%
summarise(zvit_date=min(zvit_date),
new_confirm=sum(new_confirm))
## `summarise()` ungrouping output (override with `.groups` argument)
latest <- max(area_first$zvit_date)
Крапками позначимо перший лабораторно підтверджений випадок захворювання, але скористаємось функцією fct_reorder для розташування областей в хронологічній послідовності:
(ggplot(area_first,
aes(fct_reorder(registration_area,desc(zvit_date)),
zvit_date,
size=new_confirm))
+ geom_point(color="orange")
+ scale_size_continuous(name = "Всього випадків")
+ coord_flip()
+ theme_light()
+ theme(
panel.grid.major.x = element_blank(),
panel.border = element_blank(),
axis.ticks.x = element_blank(),
legend.position = "bottom")
+ labs(title="Дата першого звіту",
subtitle="Розмір кола залежить від сумарної кількості випадків",
x="",
y=""))