covid19_ua
Аналіз "диванного експерта" відкритих даних по захворюваності на COVID19 в Україні
Project maintained by vityok Hosted on GitHub Pages — Theme by mattgraham
Розклад часових рядів
Мета: отримати тренди, сезонні коливання, випадкові відхилення різними методами.
Дані: - Сумарна кількість: - підтверджених випадків для України, - летальних випадків для України, - одужання для України, - Добова кількість підтверджених випадків для: - України - Київ
https://otexts.com/fpp3/decomposition.html
Огляд
-
Сумарна кількість:
-
підтверджених випадків для України,
-
летальних випадків для України,
-
одужання для України,
Інший метод побудови, лінії різного кольору.
library(fpp3)
library(fable)
library(tsibble)
library(feasts)
library(tidyverse)
## area_dyn <- read_csv('../covid19_by_area_type_hosp_dynamics.csv')
area_dyn <- read_csv('https://raw.github.com/VasiaPiven/covid19_ua/master/covid19_by_area_type_hosp_dynamics.csv')
daily_area_dyn <- area_dyn %>%
select(zvit_date, new_susp, new_confirm, new_death, active_confirm) %>%
group_by(zvit_date) %>%
summarise(new_susp = sum(new_susp),
new_confirm = sum(new_confirm),
new_death = sum(new_death),
active_confirm = sum(active_confirm)) %>%
mutate(susp_confirm_ratio = new_confirm / new_susp)
daily_area_dyn <- daily_area_dyn %>%
mutate(Cdate = as.Date(strptime(zvit_date, "%Y-%m-%d")))
## зібрати дані з декількох стовпчиків у новий стовпчик з назвою
## "value" (пара ключ-значення), а стара назва стовпчика буде
## збережена у стовпчику "variable"
daily_area_gath <- daily_area_dyn %>%
select(Cdate, new_susp, new_confirm, new_death, active_confirm) %>%
pivot_longer(-Cdate, names_to = "variable", values_to = "value")
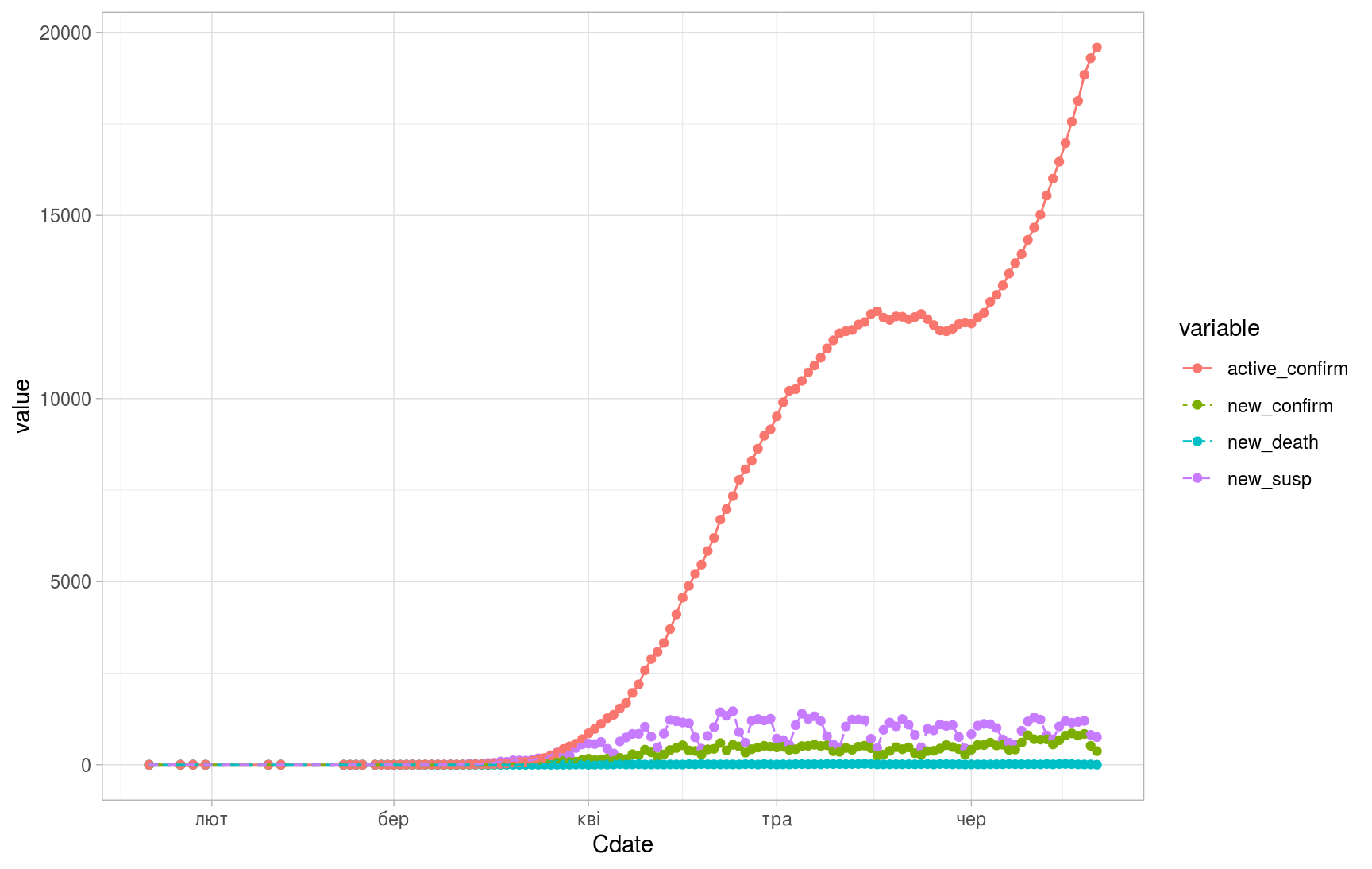
(ggplot(daily_area_gath, aes(x=Cdate,y=value))
+ geom_point(aes(color = variable))
+ geom_line(aes(color = variable, linetype = variable))
+ theme_light())
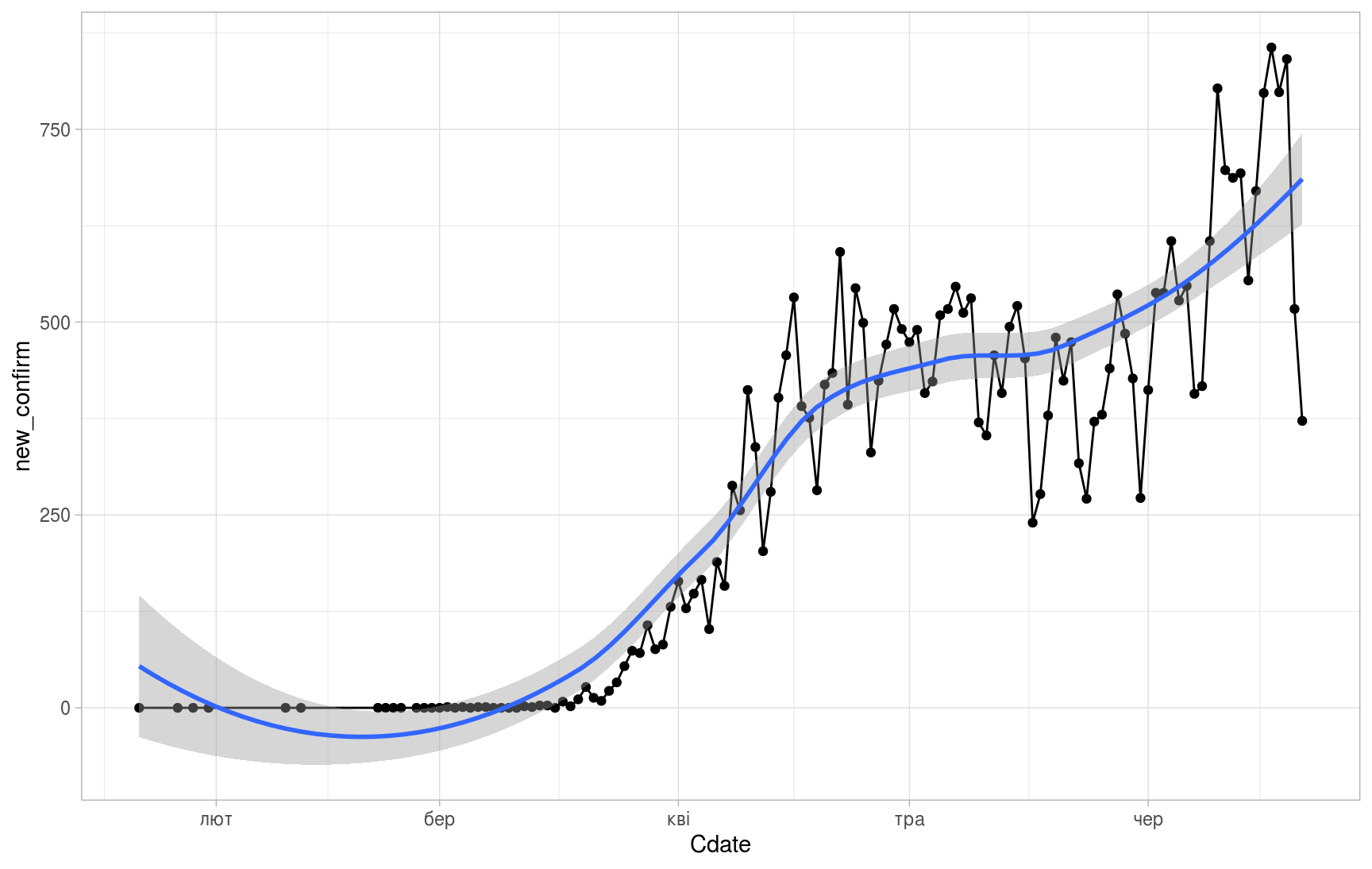
(ggplot(daily_area_dyn, aes(x=Cdate,y=new_confirm))
+ geom_point()
+ geom_line()
+ geom_smooth()
+ theme_light())
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
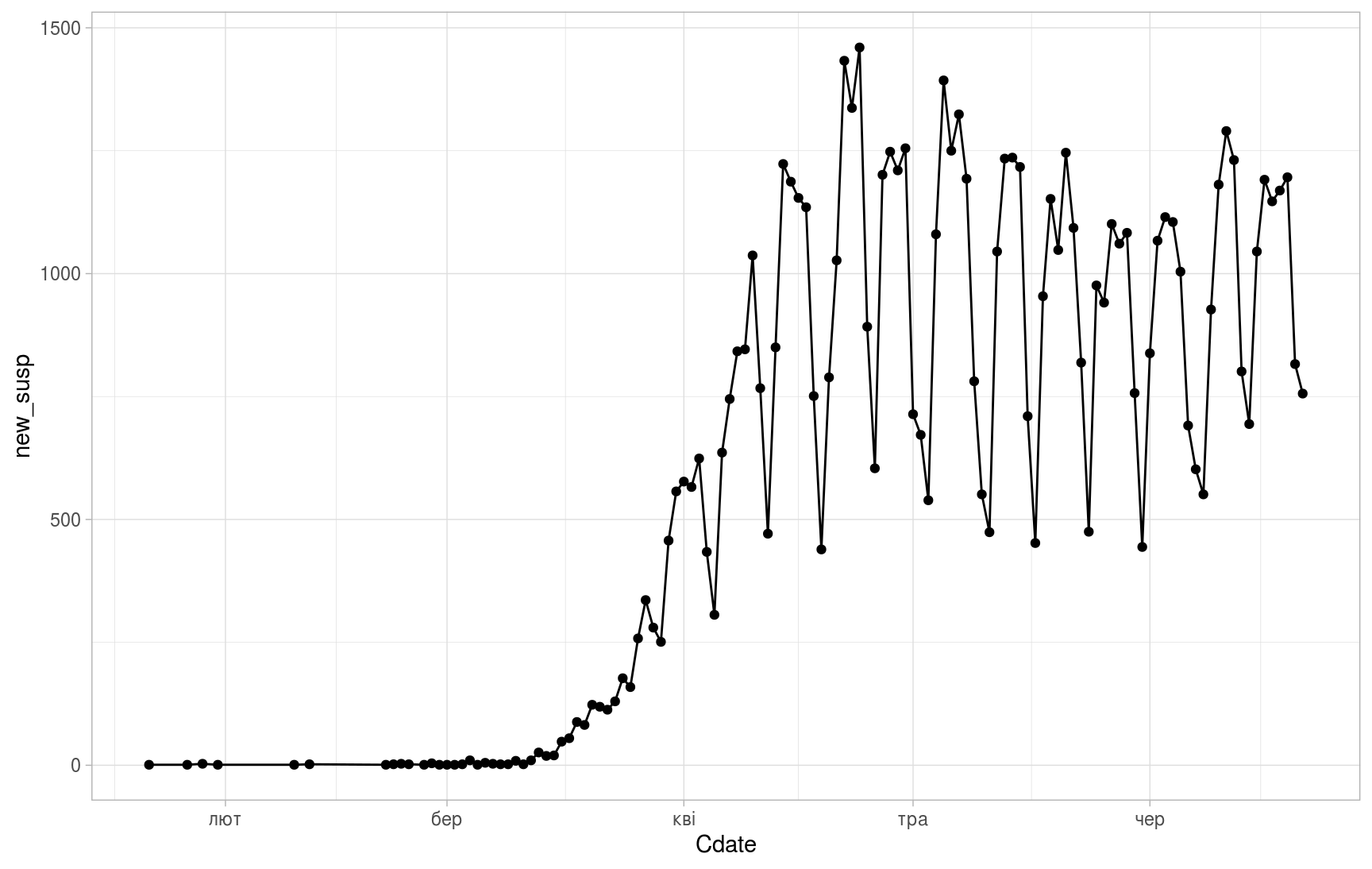
(ggplot(daily_area_dyn, aes(x=Cdate,y=new_susp))
+ geom_point()
+ geom_line()
+ theme_light())
(ggplot(daily_area_dyn, aes(x=Cdate,y=new_death))
+ geom_point()
+ geom_line()
+ theme_light())
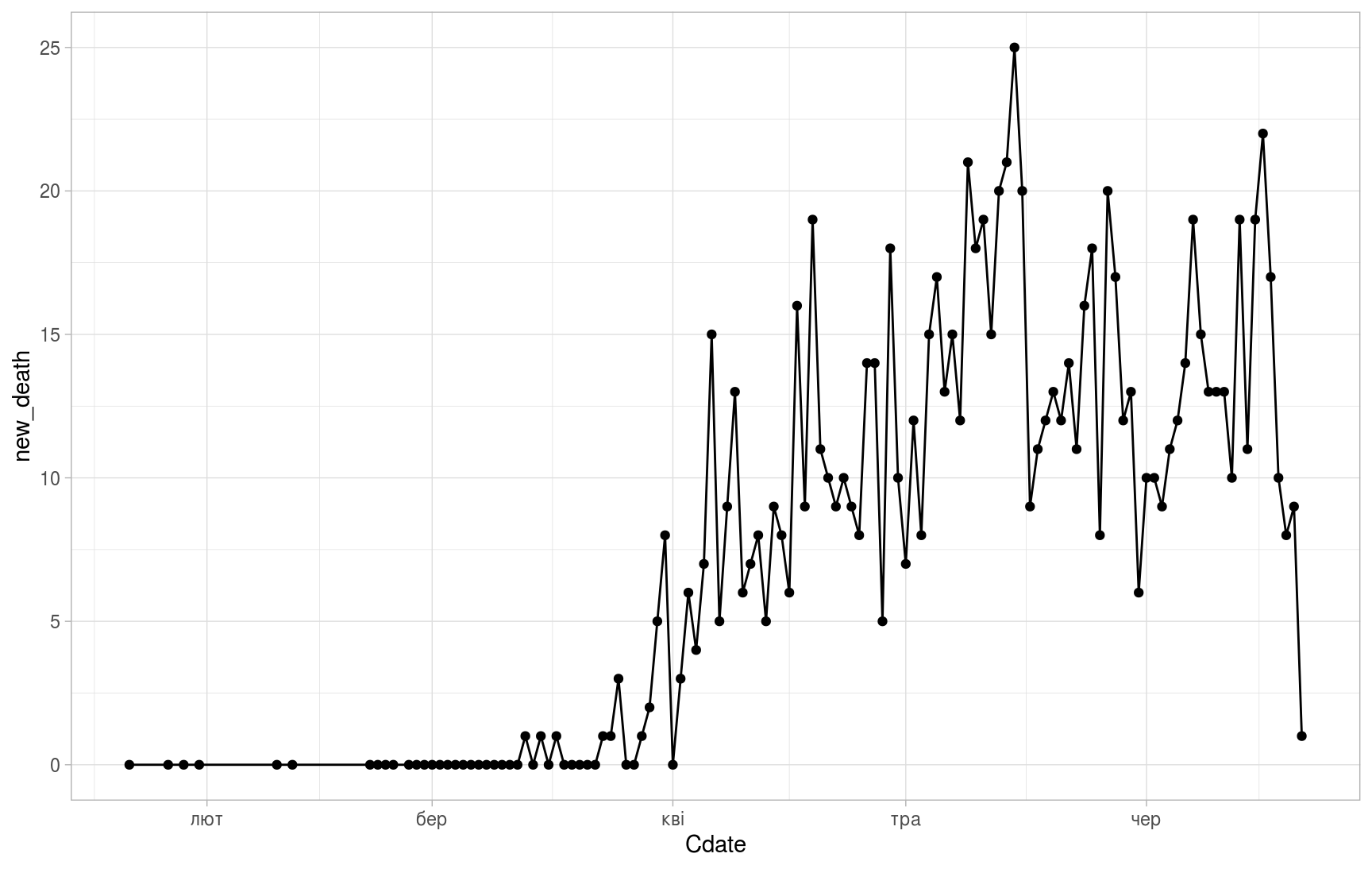
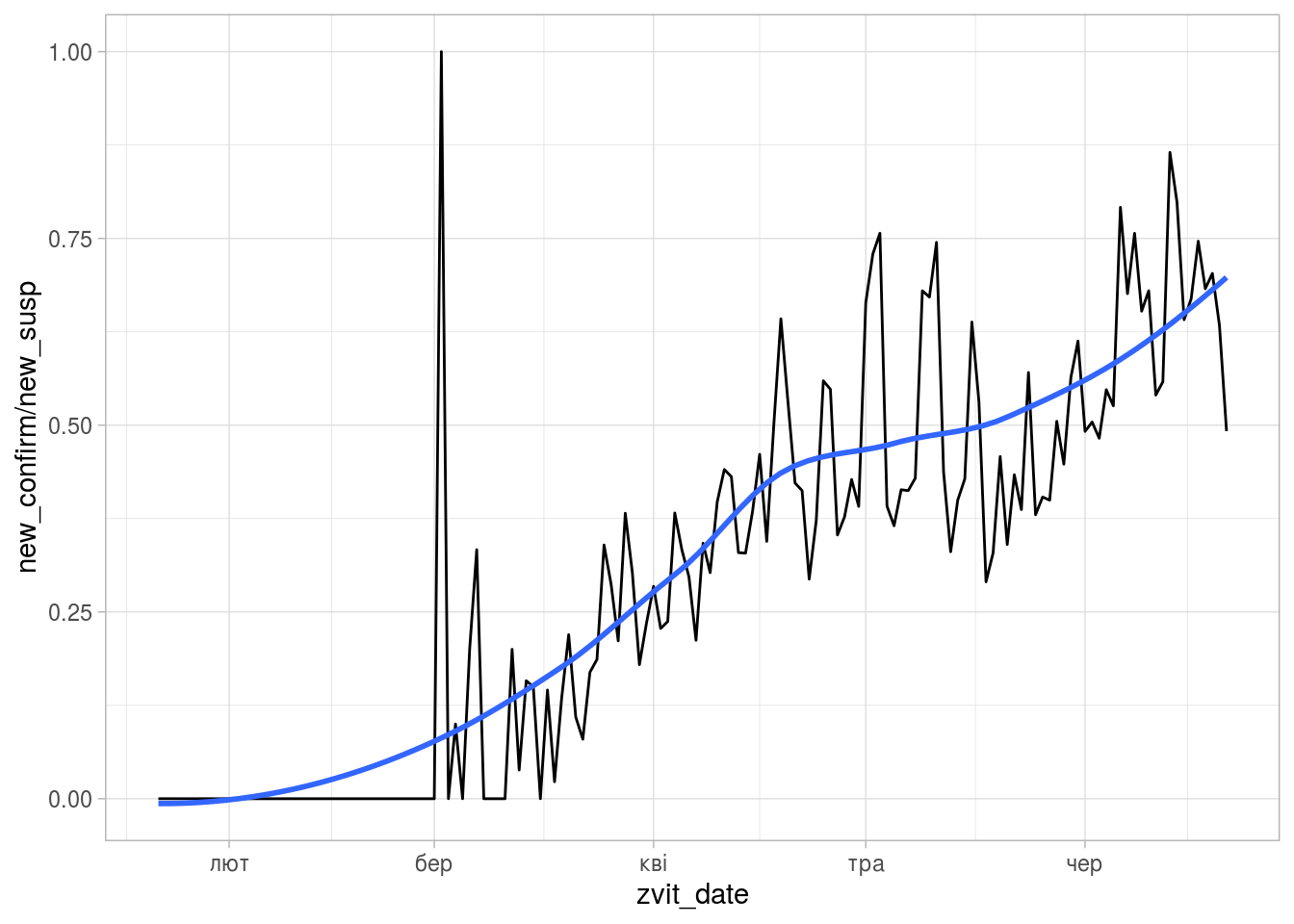
Співвідношення кількості підтверджених випадків до кількості підозр.
(ggplot(daily_area_dyn, aes(zvit_date, new_confirm/new_susp))
+ geom_line()
+ geom_smooth(se = FALSE)
+ theme_light())
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
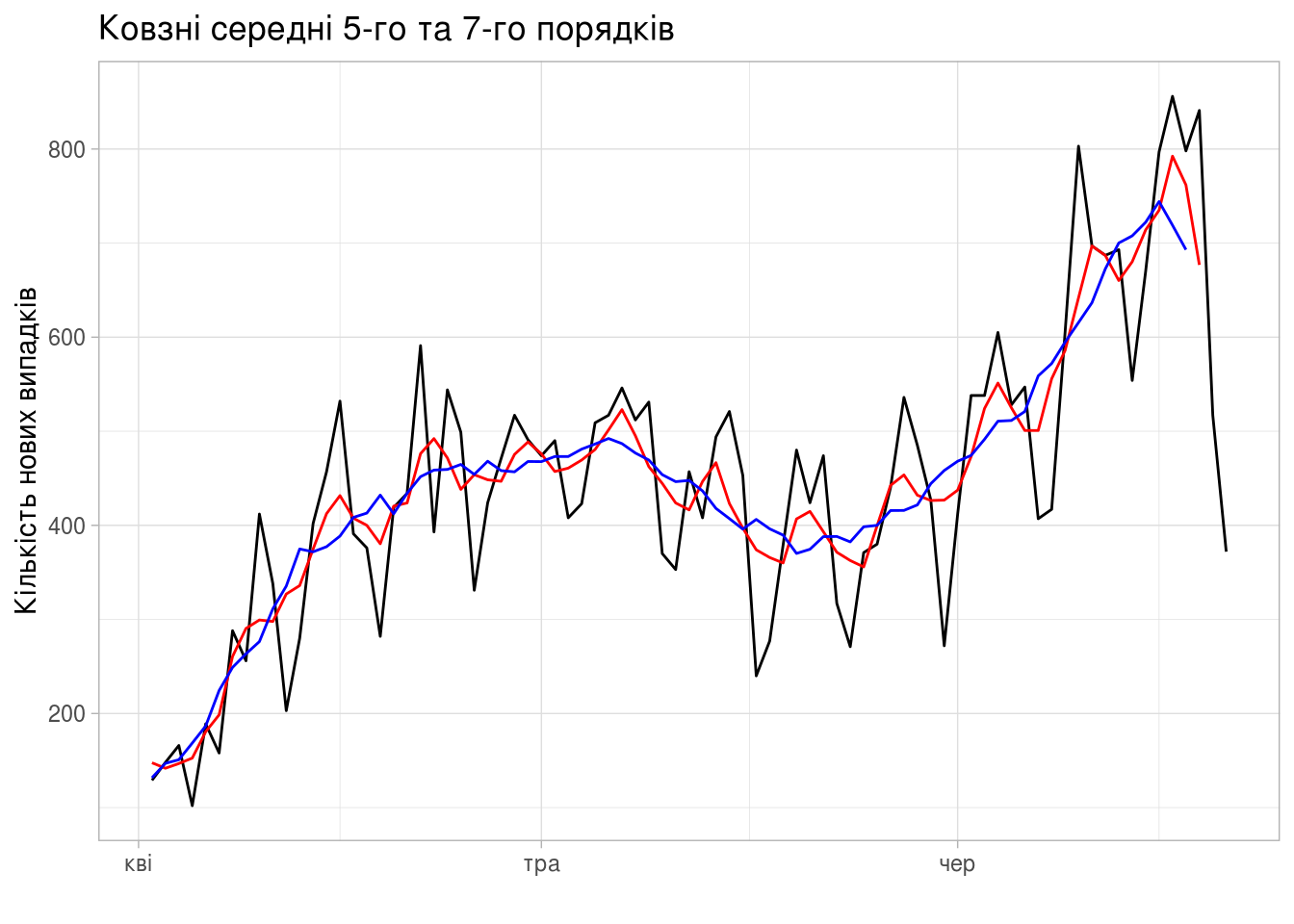
Ковзне середнє
Побудуємо ковзне середнє 5-го та 7-го порядків:
daily_area_dyn_ma <- daily_area_dyn %>%
mutate(new_confirm_5ma = slide_dbl(new_confirm, mean, .size = 5, .align = "center"),
new_confirm_7ma = slide_dbl(new_confirm, mean, .size = 7, .align = "center"),
active_confirm_7ma = slide_dbl(active_confirm, mean, .size = 7,
.align = "center"),
new_death_7ma = slide_dbl(new_death, mean, .size = 7, .align = "center")) %>%
as_tsibble(index=Cdate) %>%
filter(Cdate > as.Date('2020-04-01'))
Нових підтверджених захворювань
daily_area_dyn_ma %>%
autoplot(new_confirm) +
autolayer(daily_area_dyn_ma, new_confirm_5ma, color='red') +
autolayer(daily_area_dyn_ma, new_confirm_7ma, color='blue') +
xlab("") + ylab("Кількість нових випадків") +
ggtitle("Ковзні середні 5-го та 7-го порядків") +
guides(colour=guide_legend(title="series")) +
theme_light()
## Warning: Removed 2 row(s) containing missing values (geom_path).
## Warning: Removed 3 row(s) containing missing values (geom_path).
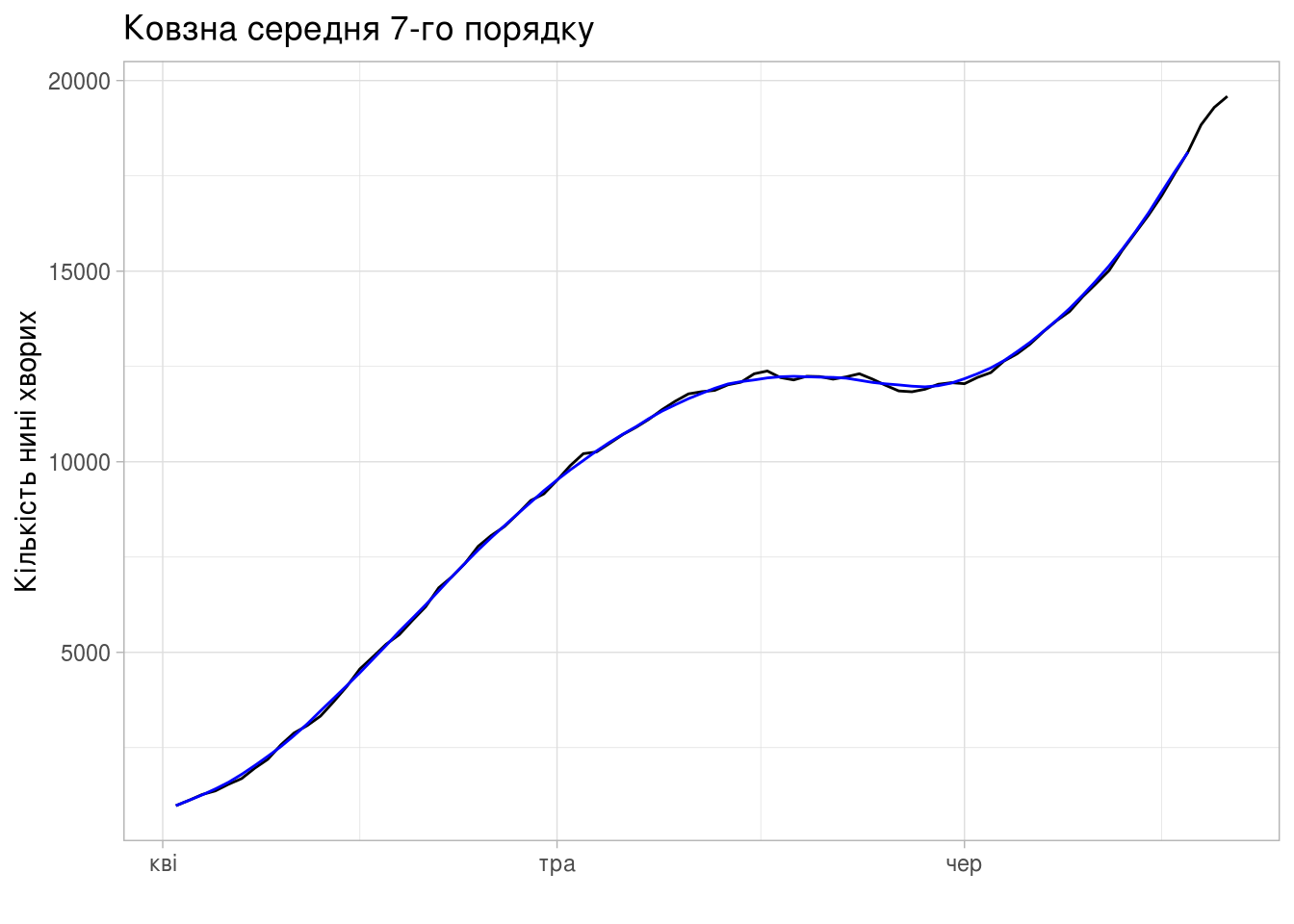
Нині хворих
daily_area_dyn_ma %>%
autoplot(active_confirm) +
autolayer(daily_area_dyn_ma, active_confirm_7ma, color='blue') +
xlab("") + ylab("Кількість нині хворих") +
ggtitle("Ковзна середня 7-го порядку") +
guides(colour=guide_legend(title="series")) +
theme_light()
## Warning: Removed 3 row(s) containing missing values (geom_path).
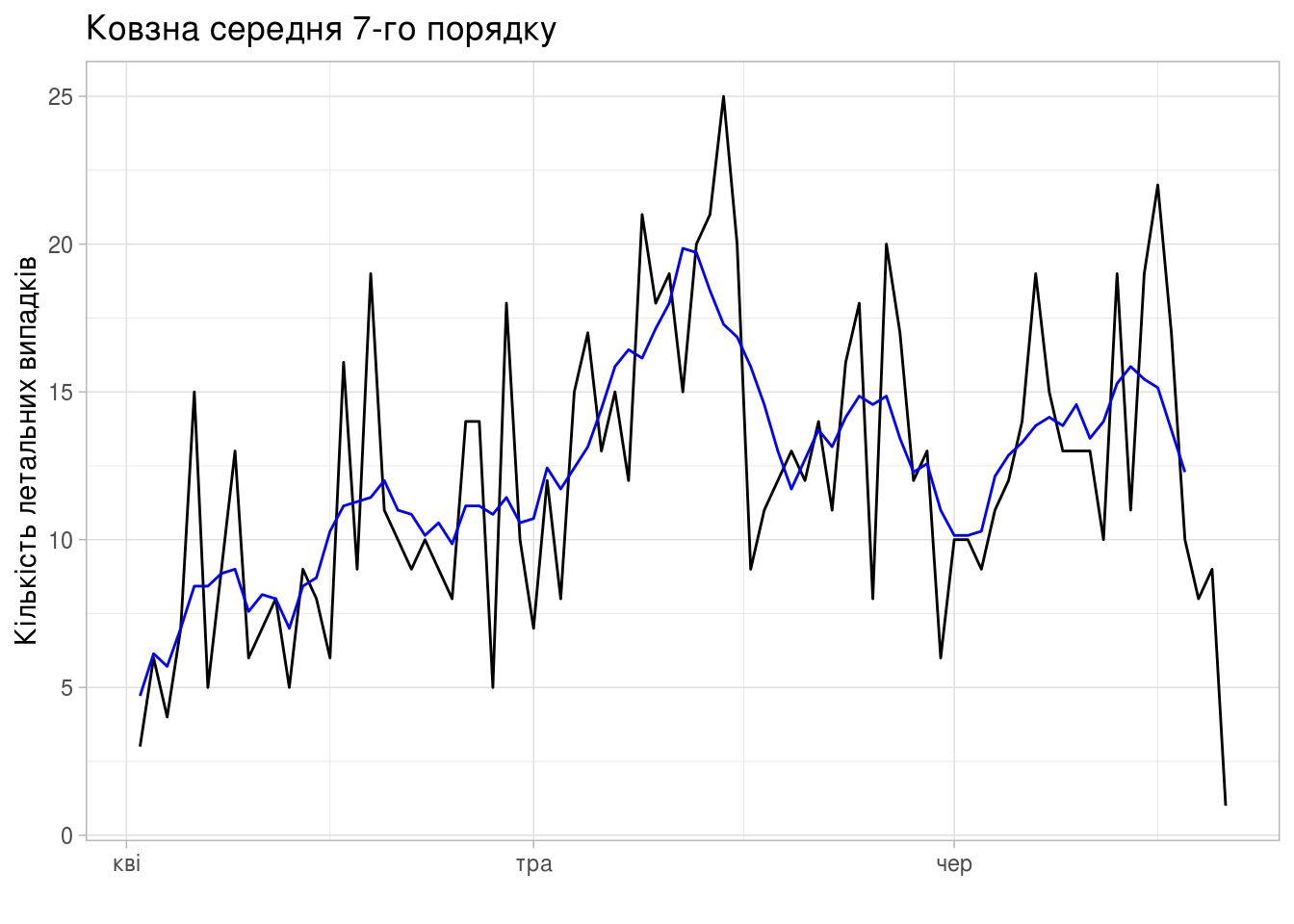
Кількість летальних випадків
daily_area_dyn_ma %>%
autoplot(new_death) +
autolayer(daily_area_dyn_ma, new_death_7ma, color='blue') +
xlab("") + ylab("Кількість летальних випадків") +
ggtitle("Ковзна середня 7-го порядку") +
guides(colour=guide_legend(title="series")) +
theme_light()
## Warning: Removed 3 row(s) containing missing values (geom_path).
Декомпозиція
Класичний розклад
Класичний розклад часового ряду на складові:
- тренд
- сезонні відхилення
- випадкові коливання (шум)
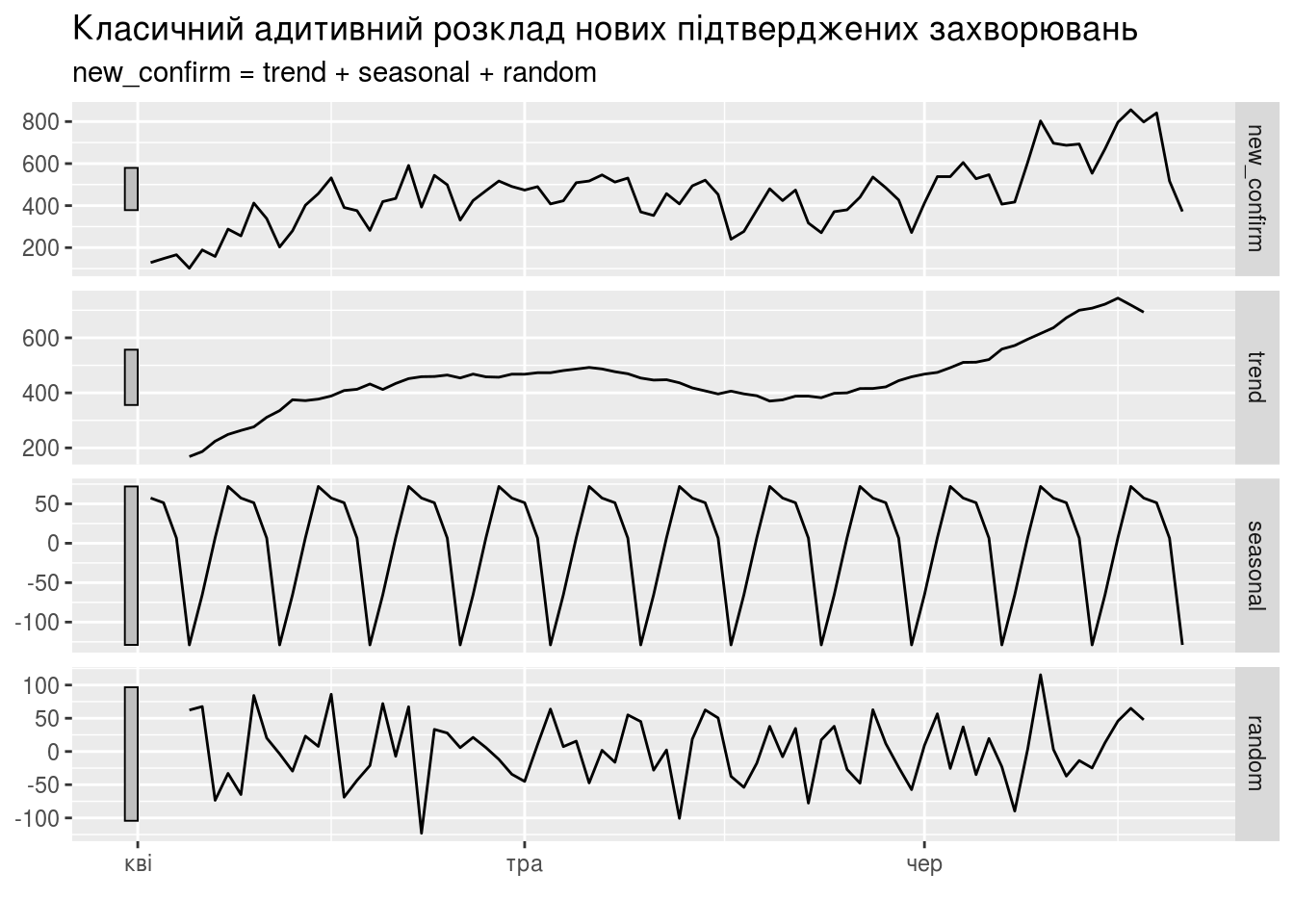
daily_area_dyn_ma %>%
model(classical_decomposition(new_confirm, type = "additive")) %>%
components() %>%
autoplot() + xlab("") +
ggtitle("Класичний адитивний розклад нових підтверджених захворювань")
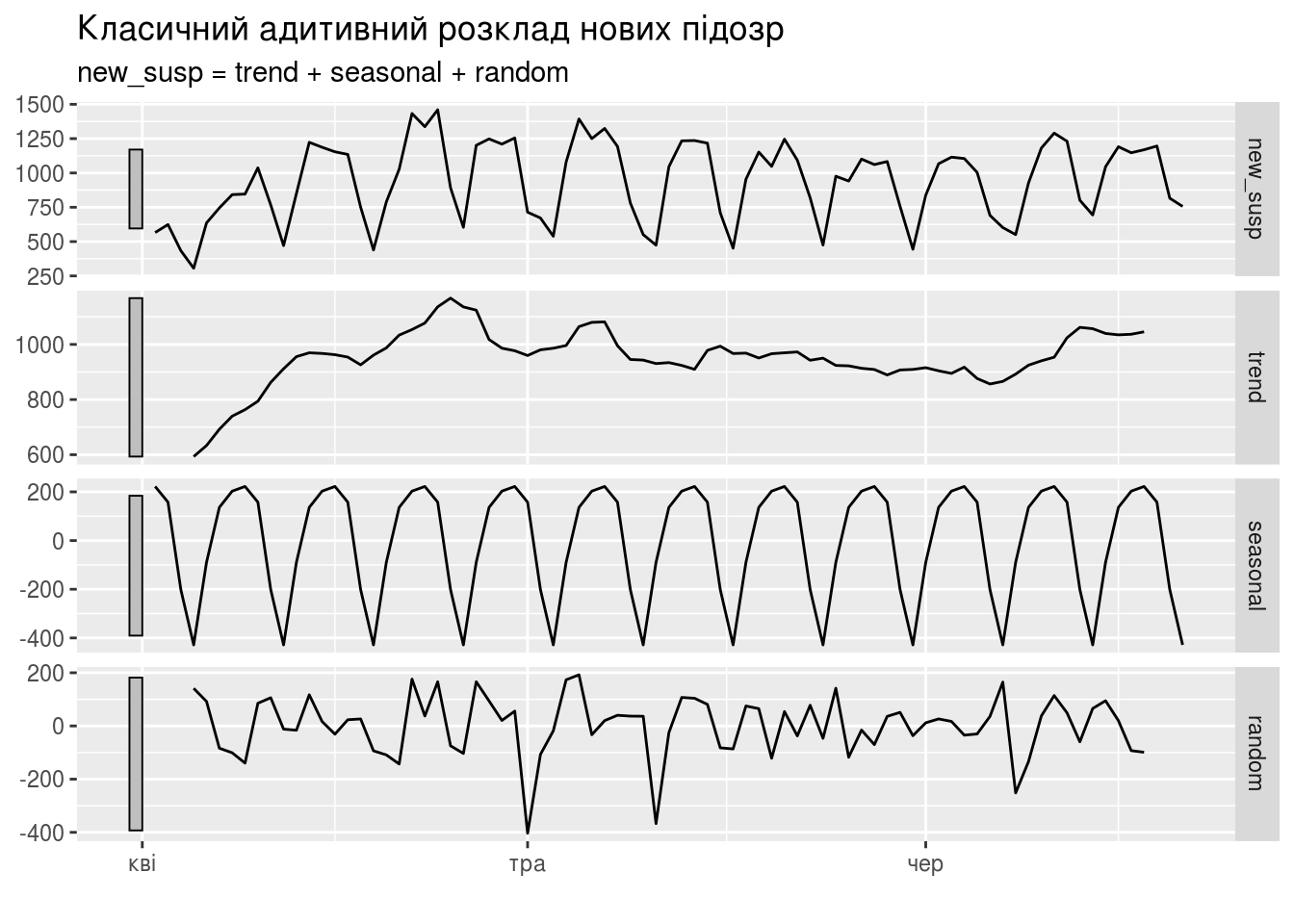
daily_area_dyn_ma %>%
model(classical_decomposition(new_susp, type = "additive")) %>%
components() %>%
autoplot() + xlab("") +
ggtitle("Класичний адитивний розклад нових підозр")
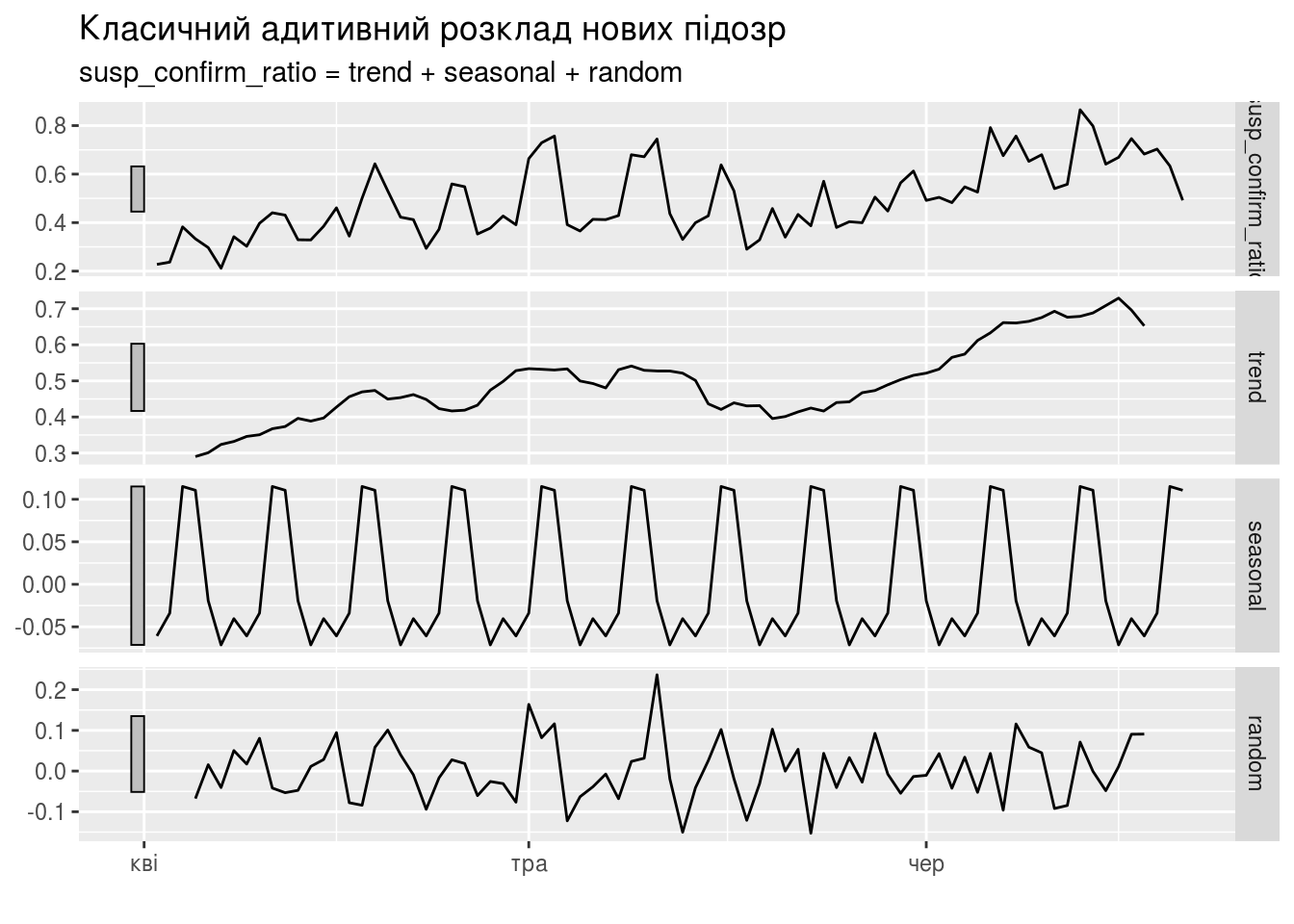
daily_area_dyn_ma %>%
model(classical_decomposition(susp_confirm_ratio, type = "additive")) %>%
components() %>%
autoplot() + xlab("") +
ggtitle("Класичний адитивний розклад нових підозр")
Методом X11
На жаль метод X11 придатний для оброблення часових рядів, періодичність яких місячна (параметр period дорівнює 12) або квартальна (параметр period дорівнює 4).
Оскільки часові ряди для захворюваності мають добовий крок, то параметр period для них становить 7.
[1] The X11 method only supports monthly (`period = 12`) and quarterly (`period = 4`) seasonal patterns
SEATS
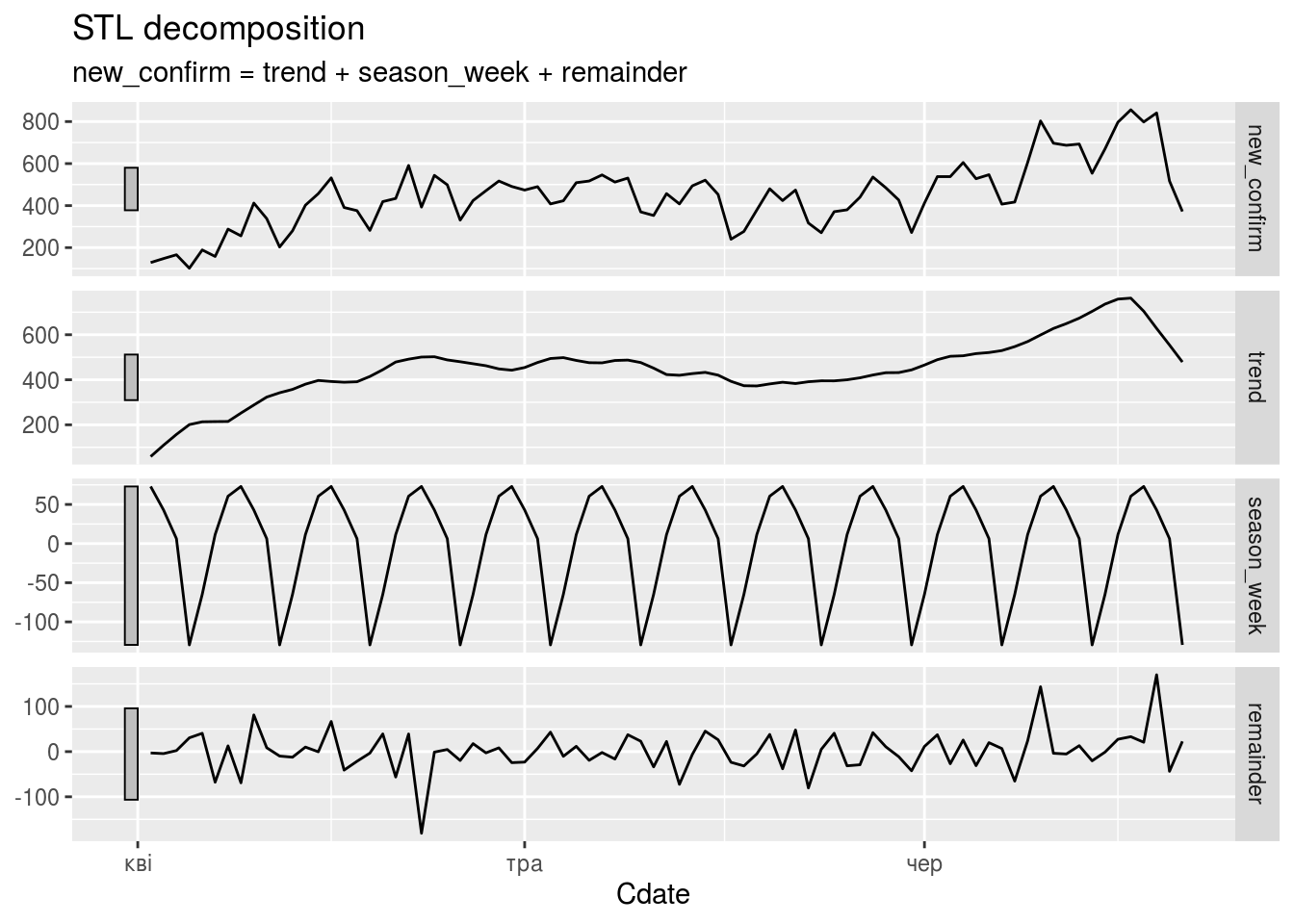
STL —
STL is a versatile and robust method for decomposing time series. STL is an acronym for “Seasonal and Trend decomposition using Loess”, while Loess is a method for estimating nonlinear relationships. The STL method was developed by Cleveland, Cleveland, McRae, & Terpenning (1990).
https://otexts.com/fpp3/stl.html
stl_dcmp <- daily_area_dyn_ma %>%
model(STL(new_confirm ~ trend(window=7) + season(window='periodic'),
robust = TRUE)) %>%
components()
autoplot(stl_dcmp)
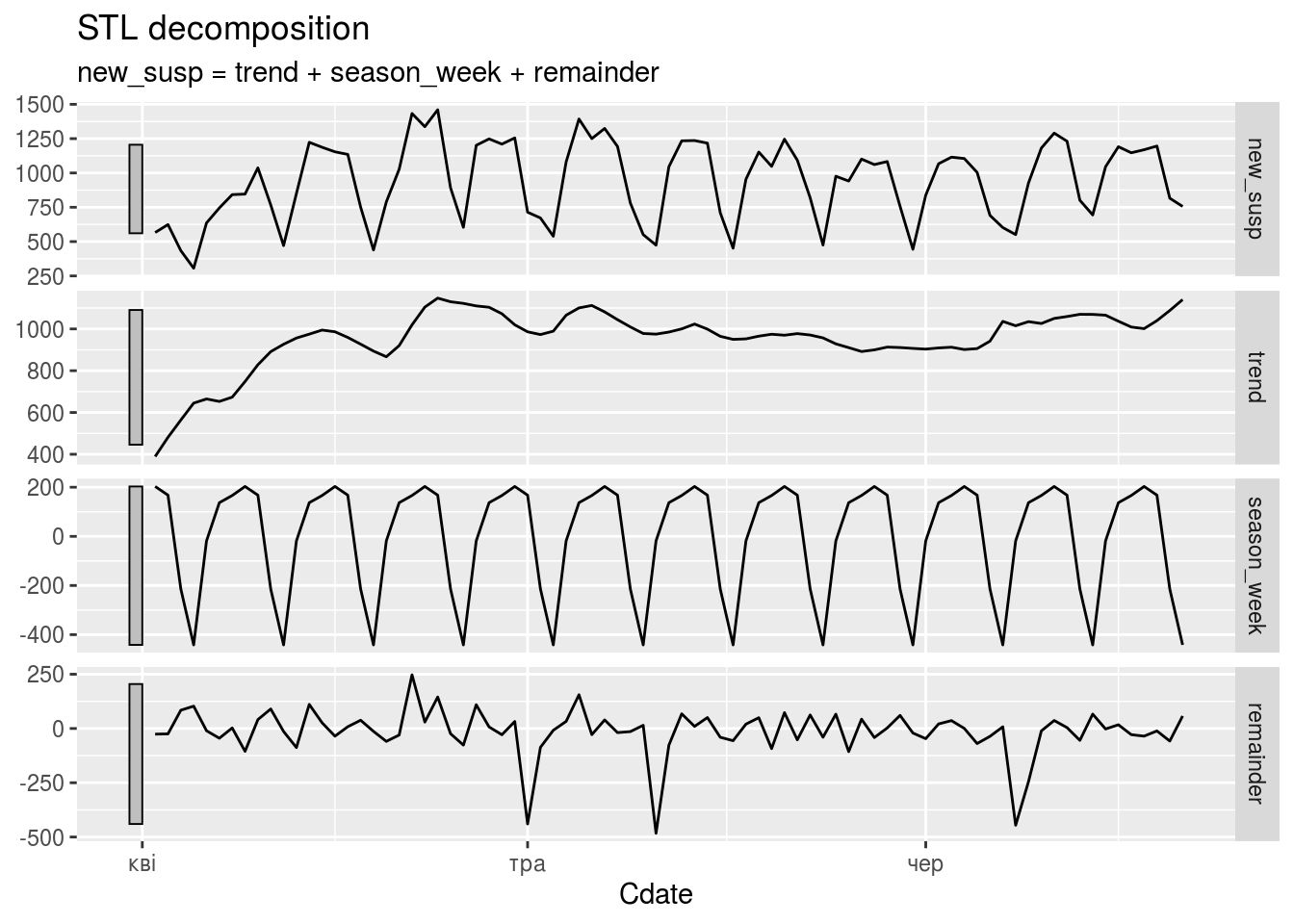
Розклад часового ряду для зареєстрованих підозр:
autoplot(daily_area_dyn_ma %>%
model(STL(new_susp ~ trend(window=7) + season(window='periodic'),
robust = TRUE)) %>%
components())
Прогнозування з декомпозицією
https://otexts.com/fpp3/forecasting-decomposition.html
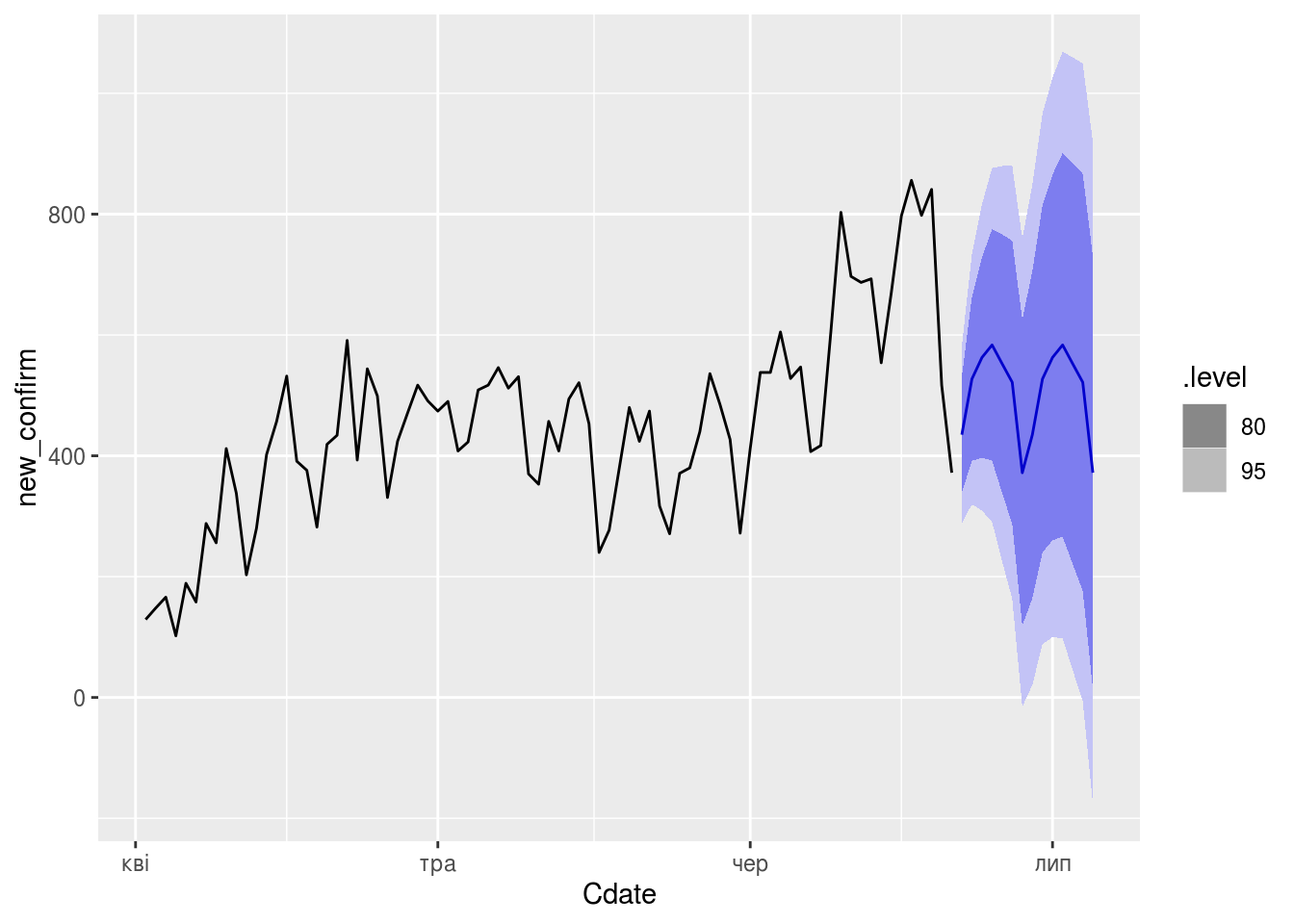
Нових захворювань
fit_dcmp_confirm <- daily_area_dyn_ma %>%
model(stlf = decomposition_model(
STL(new_confirm ~ trend(window = 7), robust = TRUE),
NAIVE(season_adjust)))
fit_dcmp_confirm %>%
forecast() %>%
autoplot(daily_area_dyn_ma)
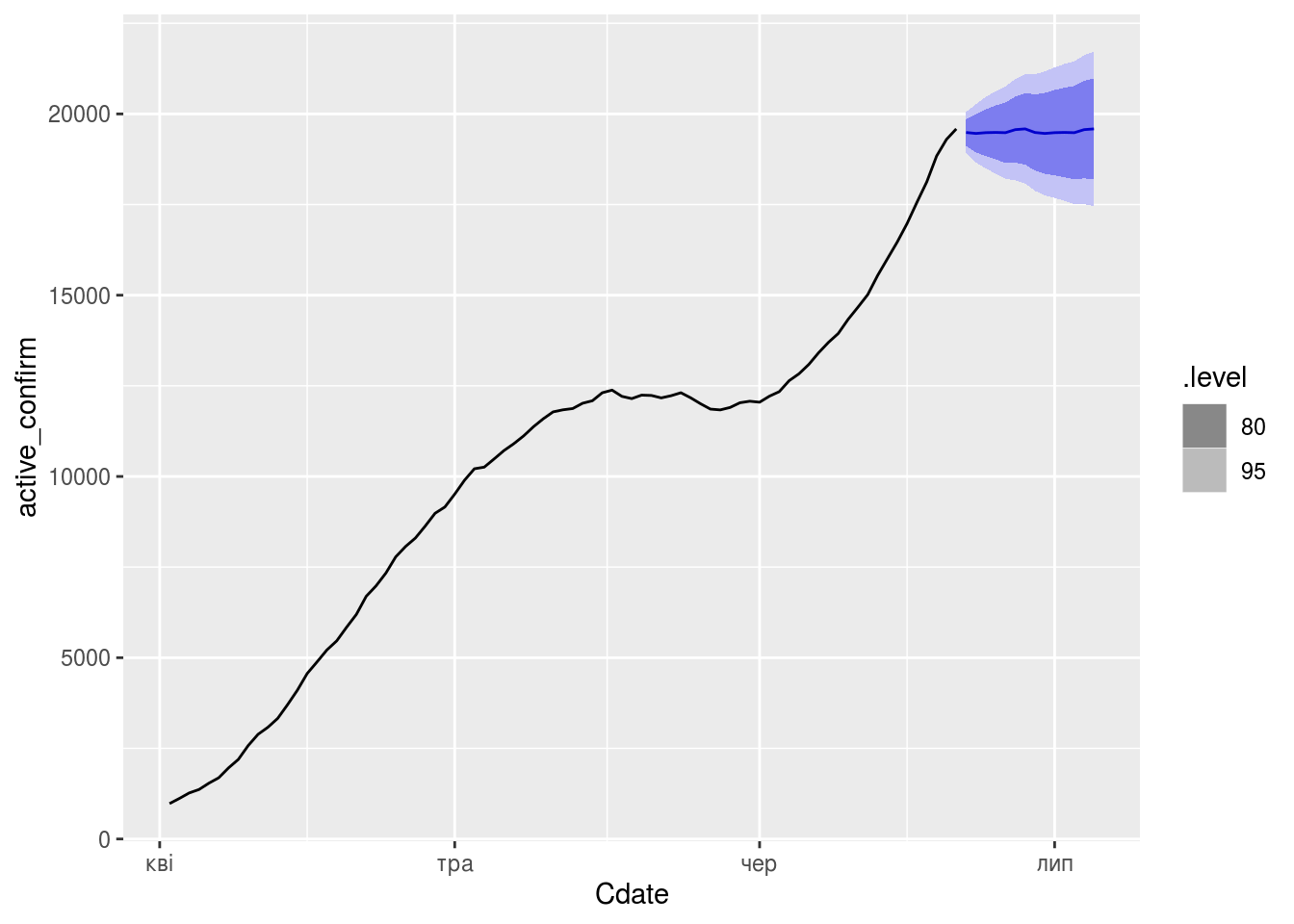
Активних
fit_dcmp_active <- daily_area_dyn_ma %>%
model(stlf = decomposition_model(
STL(active_confirm ~ trend(window = 7), robust = TRUE),
NAIVE(season_adjust)))
fit_dcmp_active %>%
forecast() %>%
autoplot(daily_area_dyn_ma)
Декомпозиція ggseas
todo: https://github.com/ellisp/ggseas
library(ggseas)